“動くDNA”が哺乳類の脳の進化過程で機能や形態の獲得に貢献するメカニズムを解明
従来、有害とされていたDNA配列が、哺乳類のゲノム進化に有益な側面を持つことが明らかに
発表のポイント
- 哺乳類の脳において、ゲノム上を“動くDNA”と呼ばれる転移因子(TE)が、進化の過程で複雑な機能の獲得に関わっていたことが近年発見されましたが、そのメカニズムは十分に解明されていませんでした。
- 本研究グループは、TEがマウスの脳神経系細胞の遺伝子制御に関わるDNA配列パターンを提供することで、細胞種の機能獲得に貢献したことを明らかにしました。また進化の過程において、特定のTE由来の遺伝子制御領域が増幅した時期を解明しました。
- この発見は、TEによる遺伝子制御領域の獲得が生物の進化の重要なトリガーになることを示しています。
概要
早稲田大学理工学術院 嘱託の関根 光太郎(せきね こうたろう)と同理工学術院総合研究所 次席研究員の小野口 真広(おのぐち まさひろ)、および同理工学術院 教授の浜田 道昭(はまだ みちあき)らの研究グループは、哺乳類の脳における特定の種類の細胞の遺伝子制御に関わるゲノム配列の一部が、「動くDNA」とも呼ばれる転移因子(Transposable element: TE) ※1によって生命進化史の中で段階的に獲得されたことを明らかにしました。
本研究成果は、Springer Nature社発行のオンラインジャーナル『Communications Biology』(論文名:Transposons contribute to the acquisition of cell type-specific cis-elements in the brain)にて、2023年6月10日(現地時間)にオンラインで掲載されました。
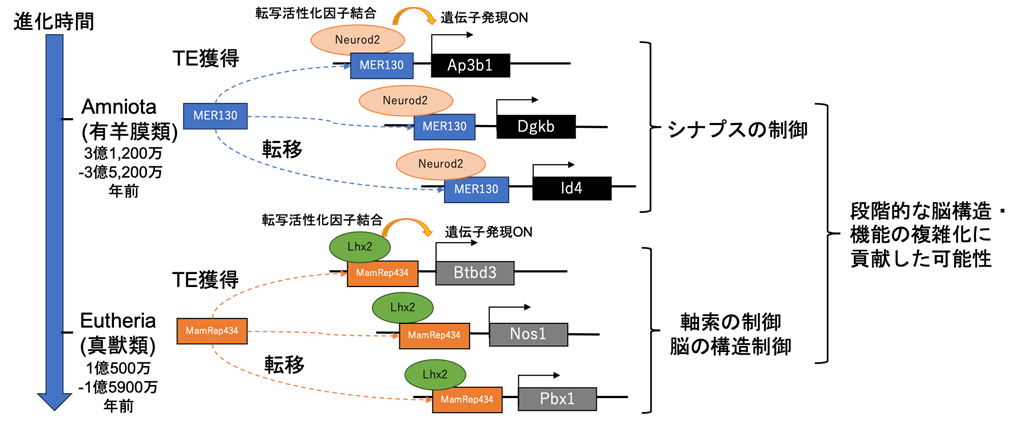
図 TE挿入による遺伝子制御ネットワークの変化と脳の形態・機能変化
(1)これまでの研究で分かっていたこと
哺乳類の脳は長い歴史の中で段階的に進化し、複雑な機能を獲得してきました。脳組織内では、神経系の細胞ごとにそれぞれ異なる遺伝子群が発現し、多様な機能・形状を持つ細胞種を生み出していることが明らかになっていますが、それらの遺伝子の発現を制御するメカニズムが進化の過程でどのように獲得されたのかについては、多くの部分が未解明なままです。
その中で近年、自身を複製・移動させることでゲノム上を動く転移因子(Transposable element: TE)と呼ばれるDNA配列が、遺伝子発現のオン/オフを決定する遺伝子制御領域として機能し、脳組織特異的な遺伝子発現パターンの獲得に関わることが発見されました。遺伝子制御領域では転写活性化因子※2が結合することで、制御先の遺伝子の発現が促されます。特定の種類のTEはその内部に転写活性化因子が結合する配列モチーフ※3を含んでおり、これらのTEがゲノム中の様々な場所に挿入されることで遺伝子制御パターンの獲得に貢献していると考えられています。
しかし、TEが組織内における細胞種の遺伝子制御機構の形成にどのように重要であるかは十分に理解されていませんでした。
(2)今回の研究で新たに実現しようとしたこと、明らかになったこと
本研究では、TEがマウスの脳組織において特定の種類の細胞の遺伝子制御に関わる配列モチーフを提供することで、転写活性化因子の結合を誘導し、細胞種の機能獲得に貢献したことを明らかにしました(図1)。特にTEの一種であるMER130※4とMamRep434※5が、それぞれNeurod2※6とLhx2※7転写活性化因子の結合する配列モチーフを内在し、グルタミン酸作動性神経前駆細胞※8において転写活性化因子の結合部位として機能することを明らかにしました。
さらに、MER130とMamRep434由来の遺伝子制御領域は、それぞれAmniota(有羊膜類)※9とEutheria(真獣類)※10の祖先において増幅されていることを発見しました(図2)。これらの結果は、TEによる遺伝子制御領域の獲得が進化の過程の複数段階で起こり、脳の複雑な機能や形態の獲得に寄与している可能性を示しています。
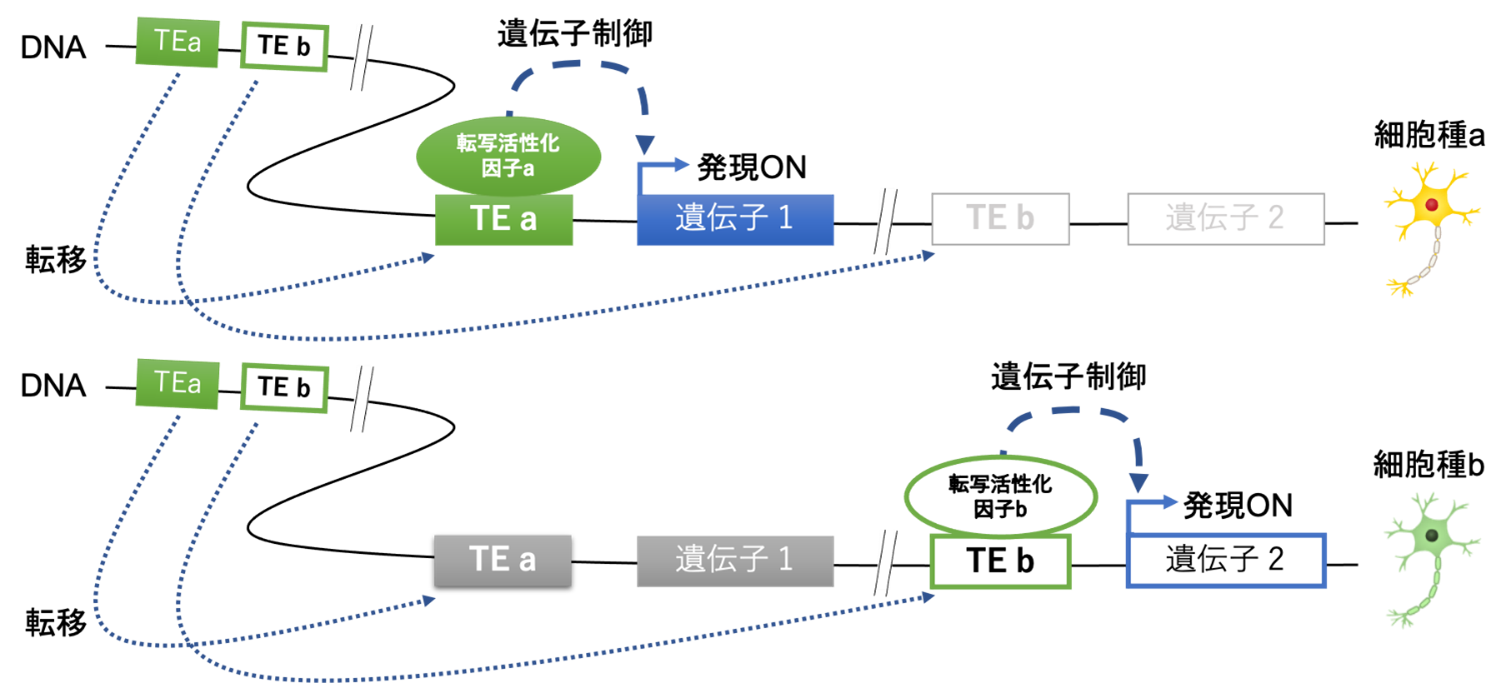
図1: 本研究の仮説(進化の過程で転移し、ゲノム中に挿入された特定の種類のTEは、転写活性化因子の結合部位として機能し、特定の種類の細胞の遺伝子発現を誘導する)
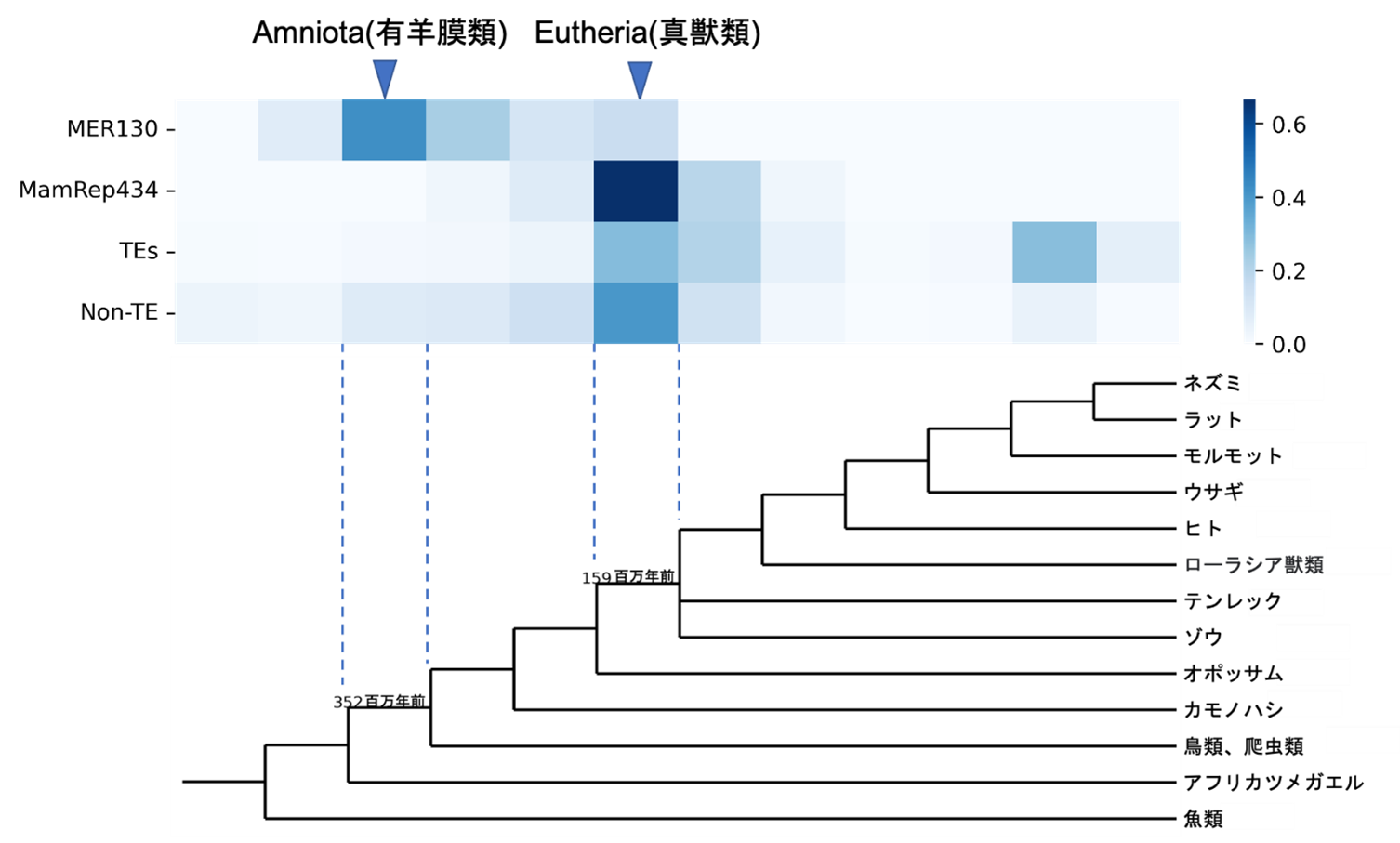
図2:進化の過程でTE由来の遺伝子制御領域を獲得した推定時期
図中のヒートマップの色の濃淡は、進化の過程ごとに獲得した遺伝子制御領域の割合(右上部縦軸の数値に対応)を示します。MER130 TE由来の遺伝子制御領域はAmniota(有羊膜類)、MamRep434 TE由来の遺伝子制御領域はEutheria(真獣類)の祖先で獲得されたことを示します。
(3)そのために新しく開発した手法
まず、1細胞ごとに遺伝子制御領域を同定することを可能とするsingle-cell ATAC-seq※11の公共データをもとに、脳組織内の特定の種類の細胞で遺伝子を制御する転写活性化因子と結合する配列モチーフを探索しました。その後で、そのモチーフを持ち、同じ種類の細胞で遺伝子制御領域として機能するTEを統計的な手法を用いて探索しました。
さらには、1細胞ごとに転写活性化因子の発現を調べられるsingle-cell RNA-seq※12の公共データや、転写活性化因子の結合領域を同定できるChIP-seq※13の公共データを組み合わせることで、特定の種類の細胞の遺伝子制御領域として機能するTEと転写活性化因子の関係を明らかにしました。
(4)研究の波及効果や社会的影響
本研究では、グルタミン酸作動性神経前駆細胞に関わる遺伝子制御配列の獲得にはTEが多大な影響を及ぼしていることがわかりました。このことは、従来考えられているような塩基置換の蓄積だけでは大規模な遺伝子制御機構の獲得が十分に説明できないことを意味しており、TEによる配列の大規模な改変が生物の進化の重要なトリガーになりうることを示しています。
TEは元来ウイルスのような外来の因子が宿主の細胞内で定着化したものであると考えられており、宿主にとって有害な側面を持つことが知られていました。本研究ではTEのゲノム進化への貢献という宿主にとって有益な側面を明らかにしました。このようなTEの複雑な機能の理解を通じて、哺乳類におけるゲノムの分子進化メカニズムと脳の進化の関係の解明につながることが期待されます。
(5)今後の課題
本研究ではマウスの成体脳を使用し、TEが成体神経再生の遺伝子制御に関わることを明らかにしました。TEは成体のみならず、成体になる前の胎児期における発生中にも遺伝子制御領域として機能することが考えられ、発生中の脳と成体脳におけるTEの機能の違いを調べることは今後の課題と考えています。また、進化の側面からTEの役割を理解するためには、マウスやヒトを含む複数の生物種の脳を比較することが不可欠と考えています。
(6)研究者のコメント
近年、分子レベルの様々な生物情報を測定する技術が飛躍的に発展しており、特に、1細胞ごとに遺伝子発現やゲノムDNAの状態を解析するシングルセル解析も様々な組織や生物種で実施されデータが蓄積されています。このような蓄積された様々なデータを組み合わせて再解析するなどの大規模データの活用が新たな生物学的知見の獲得の一つの鍵になると考えています。
(7)用語解説
※1 転移因子(Transposable element: TE)
ゲノム上で自身の配列もしくはそのコピー配列を移動させることのできるDNA配列で、“動くDNA”とも呼ばれます。ヒトのゲノム配列は約半分が転移因子で占められています。TEには転移メカニズムの異なるレトロトランスポゾン(コピー&ペースト型)とDNAトランスポゾン(カット&ペースト型)の2種類があり、その中でさらに塩基配列の特徴の異なる多数のファミリー・サブファミリーに分類されます。
※2 転写活性化因子
DNA内の遺伝子制御領域(プロモーターやエンハンサー)に結合し、対象となる遺伝子の発現を活性化させるタンパク質群を意味します。
※3 配列モチーフ
数塩基程度のDNAの塩基配列パターンを意味します。いくつかの種類の配列モチーフは、転写活性化因子が結合しやすい塩基配列の並びとして知られています。
※4 MER130
DNAトランスポゾンの一種として知られるDNA配列で、主にAmniota(有羊膜類)の祖先で獲得したことが知られています。
※5 MamRep434
DNAトランスポゾンの一種として知られるDNA配列で、主にMammalia(哺乳類)の祖先で獲得したことが知られています。
※6 Neurod2
転写活性化因子の一種で、神経細胞の分化に関わる遺伝子の発現を制御する因子として知られています。
※7 Lhx2
転写活性化因子の一種で、様々な組織の発生に関与する遺伝子の発現を制御する因子として知られています。脳では神経細胞の分化を時間・空間的に制御する役割を持つことが知られています。
※8 グルタミン酸作動性神経前駆細胞
神経前駆細胞とは、幹細胞から神経系の体細胞に分化する途中の段階にある状態の細胞を意味します。その中でグルタミン酸作動性神経前駆細胞は、興奮性ニューロンとして機能する細胞の中では最も数が多いグルタミン酸作動性神経細胞に分化する途中段階の状態の細胞を示します。
※9 Amniota(有羊膜類)
四肢動物のうち発生の初期段階に胚が羊膜を持つ生物の総称で、爬虫類・鳥類・哺乳類が挙げられます。
※10 Eutheria(真獣類)
哺乳類のうち単孔類と有袋類を除いた生物の総称です。現生の真獣類は、母体内の胎盤をもとに胎児を育てることから有胎盤類とも呼ばれます。
※11 single-cell ATAC-seq
シーケンサーを用いて1細胞ごとにDNA内の遺伝子制御領域(プロモーターやエンハンサー)を検出する方法です。
※12 single-cell RNA-seq
シーケンサーを用いて1細胞ごとにDNAから転写されるRNA(転写産物)の種類と量を網羅的に検出する方法です。
※13 ChIP-seq
DNAと特定のタンパク質の複合体を抗体で免疫沈降し、沈降したDNA断片をシーケンスすることにより、ゲノム中のどの領域が転写活性化因子などと相互作用していたのかを明らかにする手法です。
(8)論文情報
雑誌名:Communications Biology
論文名:Transposons contribute to the acquisition of cell type-specific cis-elements in the brain
執筆者名(所属機関名): 関根光太郎(早稲田大学、産業技術総合研究所)、小野口真広(早稲田大学、産業技術総合研究所)、浜田道昭(早稲田大学、産業技術総合研究所、日本医科大学)
掲載日(現地時間):2023年6月10日(土)
掲載URL:https://doi.org/10.1038/s42003-023-04989-7
DOI:10.1038/s42003-023-04989-7
(9)研究助成(外部資金による助成を受けた研究実施の場合)
- 研究費名:科学研究費助成事業 科学研究費補助金 基盤研究(A)
研究課題名:リピート要素のde novo発見に基づく長鎖ノンコーディングRNAの機能の解明
研究代表者(所属機関名):浜田 道昭(早稲田大学) - 研究費名:科学研究費助成事業 科学研究費補助金 基盤研究(A)
研究課題名:RNAを中心とした分子ネットワークに基づく生物学的相分離の俯瞰的・体系的理解
研究代表者(所属機関名):浜田 道昭(早稲田大学) - 研究費名:日本医療研究開発機構(AMED),生命科学・創薬研究支援基盤事業(BINDS)
研究課題名:1細胞/微小組織マルチオミックスのオールインワン解析による生命科学研究の支援
研究代表者(所属機関名):由良 敬(早稲田大学) - 研究費名:日本医療研究開発機構(AMED),次世代治療・診断実現のための創薬基盤技術開発事業(RNA標的創薬技術開発)
研究課題名:機能解析に基づくRNA標的創薬のための統合DBとAIシステムの構築
研究代表者:中谷 和彦(大阪大学) - 研究費名:科学研究費助成事業 新学術領域研究(研究領域提案型)
研究課題名:『学術研究支援基盤形成』先進ゲノム解析研究推進プラットフォーム
研究代表者:黒川 顕(国立遺伝学研究所) - 研究費名:科学研究費助成事業 科学研究費補助金 基盤研究(C)
研究課題名:eCLIPデータを用いた機能性RNA反復配列の探索
研究代表者名(所属機関名):小野口 真広 (早稲田大学)







