RNAアプタマー創製期間の短縮に有効な新手法RaptRankerを開発
次世代新薬開発成功率の向上とコスト低減に期待
発表のポイント
これまで次世代新薬候補RNAアプタマーの創製には多大な時間と費用を要していた
RNAアプタマーを高精度で選択でき、かつ結合に重要な情報を推定する新手法の開発に成功
次世代新薬の創製期間の短縮、適用範囲の拡大、最適化への活用などが期待されている
早稲田大学(東京都新宿区:総長 田中愛治)理工学術院の浜田道昭(はまだみちあき)教授、同大学院先進理工学研究科の石田遼我(いしだりょうが)(修士課程:研究当時)および横田彩(よこたあや)(同)ならびに、株式会社リボミック(東京都港区:代表取締役社長 中村義一)らの研究グループは、次世代の新薬の候補となるRNAアプタマー(※1)を、実験から取得される大量の配列候補から高精度で選択し、さらに、結合に重要となるモチーフ情報を推定する新手法RaptRankerを開発いたしました。また、新しく取得した実験データを用いた評価により、RaptRankerの有効性を示すことに成功いたしました。
本研究成果は、2020年6月15日(月)午前0時(協定世界時)に英国のOxford University Pressが発行するオープンアクセス科学誌「Nucleic Acids Research」で公開されました。
<これまでの研究で分かっていたこと(科学史的・歴史的な背景など)>
現在、医薬品開発においては、創薬成功率の低下、創薬プロセスの長期化と高コスト化が大きな課題となっています。これは、現在主流となっている薬(低分子化合物)では開発が困難な「膜タンパク質」などの難しい創薬ターゲットが多数残存することが一つの要因です。
一方、特定の分子に特異的かつ強く結合をするRNAは「RNAアプタマー」(以下アプタマー)と呼ばれ、次世代の新薬の要として注目されています。アプタマーの開発プロセスにおいては、まず、HT-SELEX(※2)と呼ばれる実験手法により、ターゲットとなる分子に結合するRNA配列を大量に取得し、その中から候補となるアプタマーを選択します。その後、選択した候補に対して短鎖化などの最適化を行うことにより、最終的なアプタマーを取得することが一般的な方法となっています(図1)。しかしながら、このプロセスは実験者の経験・勘に頼る部分が多く、試行錯誤的に実施され多大な時間・費用を要しており、これがアプタマー普及を妨げている一つの原因であると考えられています。

図 1 アプタマー取得プロセス
<今回の研究で新たに実現しようとしたこと、明らかになったこと>
上記の背景を受け、本研究では、HT-SELEX実験から得られる大量の配列情報から、計算機を用いて、ターゲットに対する高い親和性を持つアプタマー配列を同定する手法、さらには、その結合に重要となる配列・構造モチーフの同定する手法の開発を試みました。
今回新しく取得したHT-SELEX実験データを用いた評価により、新しく開発した手法は従来手法よりも高い精度で高親和性アプタマーの選択が可能となることが示されました。また、アプタマーの結合に重要となる配列・構造モチーフ情報の同定に成功しました(図2)。
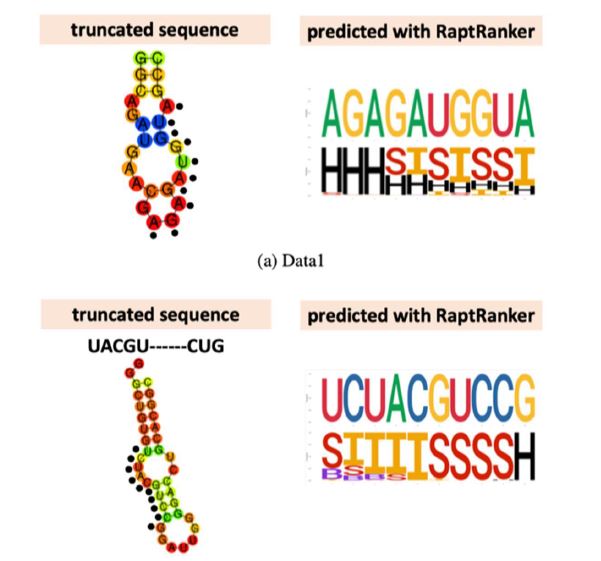
図 2 予測されたモチーフ(右)と実験により確認された二次構造(左)
<そのために新しく開発した手法>
上記を実現するために本論文では新規手法を開発し、これをRaptRankerと名付けました(図3)。RaptRankerは、HT-SELEXの全ラウンドのRNA配列情報をすべて用いて、局所的な配列・二次構造(※3)情報に基づきアプタマー候補配列のランキングを行う手法となります。さらに、結合に重要となる配列・構造モチーフの導出を行います。一般的に、アプタマーがターゲットの分子に結合する際には、アプタマー全体よりも局所的な配列・構造モチーフ(※4)が重要と考えられており、RaptRankerはこれに適した手法となっています。
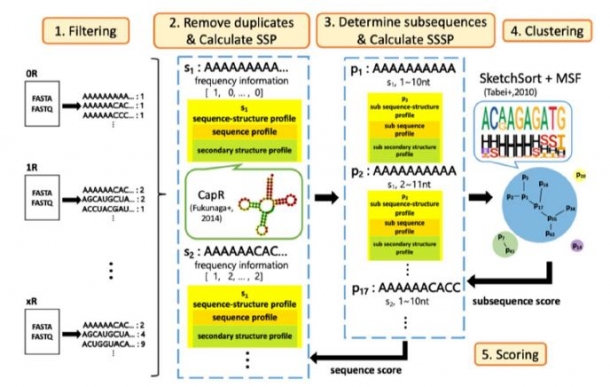
図3 新手法RaptRankerのアルゴリズム
<研究の波及効果や社会的影響>
RaptRankerを用いることで、アプタマー創製期間が現在よりも短縮することが期待されます。また、膜タンパク質など、従来手法では取得できなかった難しいターゲットに対するアプタマーが取得される可能性も秘めています。
さらに、RaptRankerにより導出されたモチーフは、アプタマーの最適化(短鎖化など)に活用可能であることが期待されます。
<今後の課題>
アプタマーデザインのさらなる効率化を目指した情報技術の開発、および、学習や評価のために必要となる実験データの取得が必要となります。
<用語解説>
※1 RNAアプタマー
- 特定の標的分子(例えば、疾患に関連するタンパク質など)に結合する20~50塩基程度の一本鎖RNAであり、塩基配列に依存した立体構造を形成し、標的の形状にフィットして結合する。RNAアプタマーは、高親和性/高特異性を有する、膜タンパク質を含む様々な分子に対して創製可能である、合成が容易である、低い免疫原性を持つなど、従来の医薬品にない数多くのメリットを有している。そのため次世代の医薬品として注目されている。
※2 HT-SELEX
- 特定のターゲット分子に強く結合するRNA配列をランダムな配列集合から何ラウンドも濃縮することにより実験的に取得する方法。各ラウンドで濃縮された配列プールを高速シークエンサーにより配列決定することにより、大量の配列情報が得られる。
※3 二次構造
- RNAの高次構造の一つで、水素結合を介した塩基対により表現される構造。ステム、ヘアピン、バルジ、内部ループ、外部ループなどの構造から構成される。二次構造は立体構造に折れたたまれる前段階であると考えられる。
※4 モチーフ
- 特定の機能(今回の場合、ターゲットに結合すること)に重要となる、短い配列や構造のこと。
<論文情報>
雑誌名:Nucleic Acids Research
論文名:RaptRanker: in silico RNA aptamer selection from HT-SELEX experiment based on local sequence and structure information
執筆者名(所属機関名):Ryoga Ishida (早稲田大学 当時)、Tatsuo Adachi(リボミック)、Aya Yokota(早稲田大学 当時)、Hidehito Yoshihara(リボミック)、Kazuteru Aoki(リボミック)、Yoshikazu Nakamura(リボミック)、Michiaki Hamada(早稲田大学、産総研・早大 生体システムビッグデータ解析 オープンイノベーションラボラトリ)
掲載予定日時(現地時間):2020年6月15日 00:05 UTC
掲載予定日時(日本時間):2020年6月15日 09:05 UTC
掲載(予定)URL:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkaa484/5855635
DOI: https://doi.org/10.1093/nar/gkaa484
<研究助成>
研究費名:JST戦略的創造研究推進事業(CREST)
研究課題名:人工知能技術を用いた革新的アプタマー創薬システムの開発
研究代表者名(所属機関名):浜田道昭(早稲田大学)







