狙った場所にドナーDNAを挿入するノックイン新手法(BiPoD)を開発
6%(通常法)から90%超(新手法)に 正確性と効率が大幅に向上
発表のポイント
- 従来のノックイン手法は効率の低さと、意図しないドナーDNAの挿入の発生によるゲノム破壊などの課題を抱えていた。
- DNA修復の仕組みを一時的にブロックすることで、部分的な挿入によるゲノム破壊が起こらない正確なノックイン手法を確立した。
- 新手法により90%超の高い確率で2組の染色体両方に同時にノックインさせることに成功した。
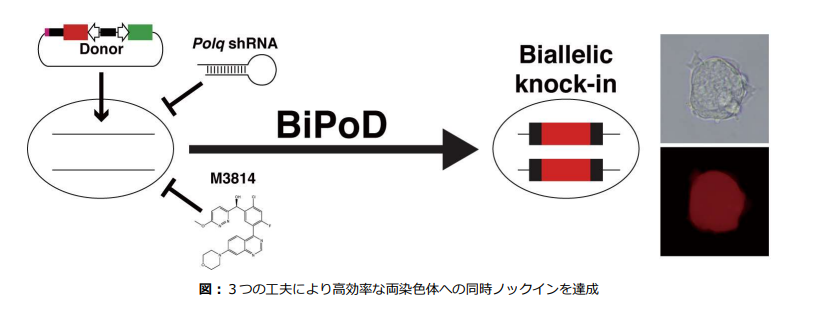
早稲田大学理工学術院の新井大祐(あらいだいすけ)講師および中尾洋一(なかおよういち)教授らの研究グループは、ゲノムの狙った場所にドナーDNAを挿入する、正確性と効率性が高い新たな相同組換えノックイン手法となるBiPoD(Biallelic knock-in assisted by Pol θ and DNA-PK inhibition)を開発しました。
従来のノックイン手法は効率性の低さに課題を抱え、研究の足枷となっていました。また、この手法では、ドナーDNAの一部分だけが頻繁に挿入するため、これにより元々の遺伝子を破壊したり検出が困難になったりすることが判りました。
そこで今回、本研究グループは、従来の手法よりも相同組換えの効率を高めつつ、それ以外の仕組みによるドナーDNAの挿入をほぼ完全に抑えられる新たな手法を確立しました。これにより、部分的挿入などによるゲノムの破壊が起こらない正確で安全なノックインが可能になりました。またこの新手法は90%超の高い確率で2組の染色体両方に同時にノックインさせることに成功しました。正確性と効率が高まった新手法は、マウスES細胞を用いた研究を加速させることが可能です。さらに、簡便な手法で特別な機器や高価な試薬を必要としないために、導入が容易なため、相同組換えノックイン手法の新たなスタンダードとして広く普及することが期待できます。
本研究成果は、Springer Nature社『Scientific Reports』のオンライン版に2021年9月13日(現地時間)に掲載されました。
【論文情報】
雑誌名:Scientific Reports
論文名:Efficient biallelic knock-in in mouse embryonic stem cells by in vivo-linearization of donor and transient inhibition of DNA polymerase θ/DNA-PK
DOI:10.1038/s41598-021-97579-8
1.これまでの研究で分かっていたこと(科学史的・歴史的な背景など)
生命の設計図であるゲノムDNAの中にある特定の遺伝子を破壊したり、外来のドナーDNA※1を狙った場所に挿入(ノックイン)したりする技術は基礎研究に欠かせないものです。また、遺伝子治療や細胞医療といった最先端医療への活用も期待されています。
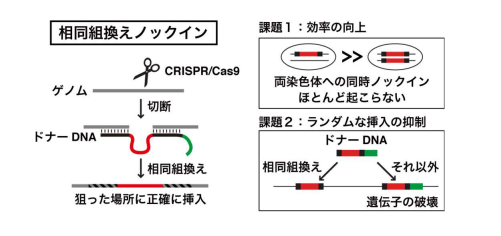
CRISPR/Cas9システム※2を細胞が元々持っているDNA修復の仕組みである相同組換え※3に組み合わせたノックインは、主要なゲノム編集技術として現在も世界中で使われています。人間の細胞の中には遺伝子のセットである染色体が2組存在しており、同じ遺伝子が2つずつ存在しています。よってノックインにより遺伝子の働きを操作する場合、ドナーDNAを両方の染色体に挿入する必要があります。しかし相同組換えにより2組の染色体に同時にノックインされる確率は数%未満と非常に低く、研究を進める上での足枷となっていました。
また従来の手法では、相同組換えノックインと同時に、標的と無関係な場所へのドナーDNAのランダムな挿入が発生してしまいます。ランダムな挿入により挿入箇所に元々あった遺伝子などが破壊されると、細胞に予期せぬ影響を及ぼします。これはノックインを臨床に応用するにあたり必ず解決しなければならない問題でした。
2.今回の研究で新たに実現しようとしたこと、明らかになったこと
本研究では、マウスのES細胞を対象に、正確性と効率を高めた新たな相同組換えノックイン手法の確立に取り組みました。
これまでは相同組換えノックインを行う場合、ドナーDNAに複数のマーカー遺伝子※4を配置することで、正しくノックインされた細胞と、相同組換え以外の仕組みによりドナーDNA全体がランダムに挿入された細胞を区別し、後者の細胞を除外するという戦略がとられてきました。しかし本研究を進める過程で、通常の手法ではドナーDNAの一部分だけのランダムな挿入が頻繁に発生することがわかりました。これは挿入箇所に元々ある遺伝子を破壊してしまう上に、挿入の仕方によってはマーカー遺伝子による検出が不可能になります。よって後からマーカー遺伝子によって除外するのではなく、最初から部分的挿入が起こらないような手法が必要と考えられました。
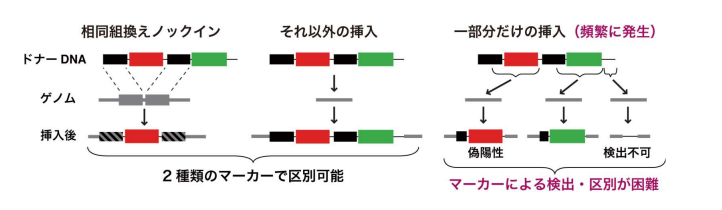
部分的挿入は相同組換え以外のDNA修復の仕組み(非相同末端結合、マイクロホモロジー媒介末端結合)を介して起こります。これらの仕組みを一時的にブロックすることで特異的な相同組換えノックインが達成できないか検討しました。その結果、ドナーDNAの構造の工夫、小分子化合物M3814※5による非相同末端結合の阻害、Polq遺伝子※6に対するshRNA※7によるマイクロホモロジー媒介末端結合の阻害を組み合わせることで、通常法よりも相同組換えの効率を高めつつ、それ以外の仕組みによるドナーDNAの挿入をほぼ完全に抑えられる手法を確立しました。
本手法によって、部分的挿入などによるゲノムの破壊が起こらない正確なノックインが可能になりました。これにより細胞への予期せぬ悪影響を防ぐことができ、遺伝子治療や細胞医療にノックインを用いる際の安全性が大幅に向上しました。さらに予想外の発見として、この手法では90%超の確率で2組の染色体両方に同時にノックインが起こることがわかりました(通常法では6%)。相同組換えノックインの改良法についてはこれまでに多数の報告がありますが、それらと比べても両染色体への同時ノックイン効率は遥かに高いものでした。そこで本手法を新たな相同組換えノックイン手法としてBiPoD(Biallelic knock-in assisted by Pol θ and DNA-PK inhibition)と命名しました。
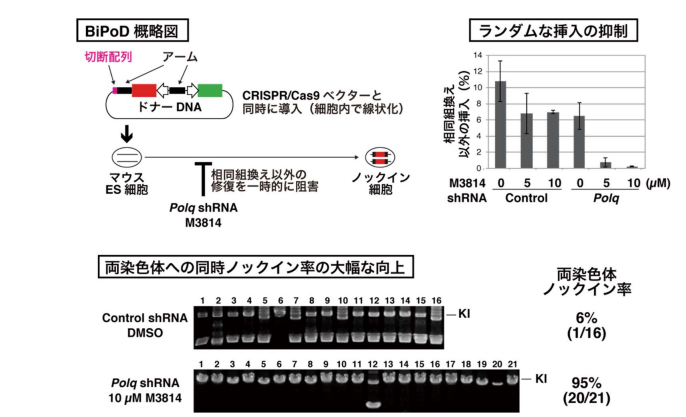
図左上:BiPoDの概略図。
図右上:ランダムな挿入の発生率。M3814とPolq shRNAを組み合わせることでランダム挿入が大幅に抑えられている。
図下:ゲノムPCR法によるノックイン状況の確認。通常法16種(上段)、BiPoD条件21種(下段)のクローンを単離、解析している。各クローンについて、上側のバンドがノックインされた染色体、下側のバンドがノックインされなかった染色体を示しており、上側だけにバンドがあるクローンは両染色体にノックインが起こっている。BiPoDにより21クローン中20クローン(95%)が両染色体ノックインとなり、通常法(16クローン中1クローン、6%)よりも両染色体ノックイン率が大幅に向上した。
またBiPoDを用いることにより、通常法では不可能だった2箇所の標的への両染色体同時(計4箇所)ノックインも高効率に達成できることも示されました。
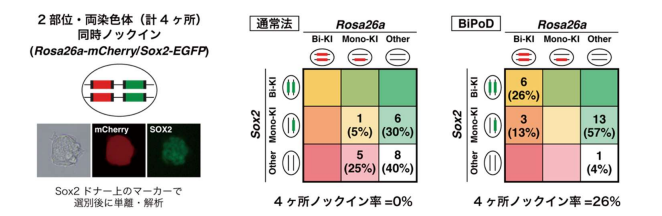
図:2部位(Rosa26a、Sox2)への同時ノックインを行い、ゲノムPCR法によりクローンの解析を行なった。各部位について、両染色体(Bi-KI)、片方の染色体(Mono-KI)、ノックインなし(Other)のいずれであるかを解析し、クローン数とパーセンテージを示した。通常法では両染色体ノックインはどちらの部位にも起こっていなかったが、BiPoDでは9クローン(39%)がRosa26aに両染色体ノックインされており、うち6クローン(26%)はSox2にも両染色体ノックインが確認され、4箇所同時にノックインしていることが示された。
3.研究の波及効果や社会的影響
高効率かつ特異性の高い手法であるBiPoDはマーカー遺伝子を使わずに両染色体ノックイン細胞を樹立することや、ワンステップで複数のノックインを施すことを可能とし、マウスES細胞を用いた研究を大幅に加速させるポテンシャルを有しています。また非常に簡便な手法で特別な機器や高価な試薬も必要としないので、研究室への導入も容易です。相同組換えノックイン手法の新たなスタンダードとして広く普及していくことを期待しています。
4.今後の課題
BiPoDがマウスES細胞以外の細胞、特にヒトの細胞に対しても有効かどうかを確認する必要があります。そのままの手法で十分な効果が見られない場合、何らかの微修正を加えていく必要があるでしょう。また、shRNAによる遺伝子ノックダウンを何らかの薬剤に置き換えることができれば、作業がさらに簡易なものになります。シンプルな手法なだけに、改良の余地はまだまだ残されていると考えています。両染色体へのノックイン効率が劇的に上昇するメカニズムの詳細についても、今後調べていく予定です。
5.研究者のコメント
元々は自身の日々の実験を効率化するために始めた日曜大工のようなテーマでしたが、予想を上回る優れた効果が認められたため、世の中に広く知られてほしいと考え、論文にまとめて発表しました。この手法が自分の研究だけでなく、国内外の多くの研究グループに活用され、新たな科学的発見の一助となることを願っています。
6.用語解説
※1 ドナーDNA
- 細胞外からゲノムに挿入されるDNA配列。外来遺伝子の挿入や元々ある遺伝子に人工的な配列を付加するなど、目的に応じて自由に設計することができる。
※2 CRISPR/Cas9システム
- 2012年に開発された、ガイドRNAと呼ばれるRNAとCas9というタンパク質を細胞に導入することで、ゲノムDNAの狙った場所だけを特異的に切断するゲノム編集技術。
※3 相同組換え
- 切断されたゲノムDNAを修復する仕組みの一つで、切断箇所の両側と同じ配列(アーム)を持つDNAを鋳型として修復を行う。修復の鋳型としてドナーDNAが使われると、アームに挟まれたDNA配列も一緒に挿入される。CRISPR/Cas9システムによりゲノムDNAを切断することで、狙った場所に効率的に相同組換えを引き起こすことができる。
※4 マーカー遺伝子
- ドナーDNAがゲノムに挿入されたことを示す目印となる遺伝子。蛍光タンパク質や抗生物質に対する耐性物質を作る遺伝子が用いられる。フローサイトメトリーや抗生物質処理によりマーカー遺伝子を持つ細胞だけを集めることができる。
※5 M3814
- DNA-PKというタンパク質の働きを阻害することにより、非相同末端結合をブロックする薬剤。
※6 Polq遺伝子
- マイクロホモロジー媒介末端結合に必須の因子であるDNA polymerase θ(Pol θ)をコードしている遺伝子。
※7 shRNA
- 細胞に導入したプラスミドDNAから転写され、ヘアピン型の構造をとった短いRNAのことで、RNA干渉により標的遺伝子の働きを一時的に抑制することができる。
7.論文情報
雑誌名:Scientific Reports
論文名:Efficient biallelic knock-in in mouse embryonic stem cells by in vivo-linearization of donor and transient inhibition of DNA polymerase θ/DNA-PK
執筆者名(所属機関名):新井大祐1*、中尾洋一1,2(1:早稲田大学 先進理工学部 化学・生命化学科、2:早稲田大学 理工学術院総合研究所、*責任著者)
掲載日時(現地時間):2021年9月13日10:00
掲載日時(日本時間):2021年9月13日19:00
掲載URL:www.nature.com/articles/s41598-021-97579-8
DOI:10.1038/s41598-021-97579-8
8.研究助成(外部資金による助成を受けた研究実施の場合)
本研究は日本学術振興会(JSPS)科学研究費助成事業の助成を受けて行われました。







