発表のポイント
- 分裂酵母細胞が休眠から目覚める際の遺伝子の発現状態変化について解明した。
- その結果、ヒストンH3遺伝子の変動が休眠打破に重要な役割を果たすことを明らかにした。
- 今後新たな制がん標的遺伝子の特定や、がん細胞の増殖を抑える創薬への道筋が期待される。
早稲田大学理工学術院の佐藤 政充(さとう まさみつ)教授および同大大学院先進理工学研究科博士2年の露崎 隼(つゆざき はやと)氏の研究グループは、同大理工学術院の竹山 春子(たけやま はるこ)教授および細川 正人(ほそかわ まさひと)次席研究員の研究グループ、産総研・早大 生体システムビッグデータ解析オープンイノベーションラボラトリ(CBBD-OIL)※1との共同研究により、シングルセル解析技術※2を改良することで、分裂酵母※3細胞が休眠から目覚める際の遺伝子の発現状態の変化について解明しました。その結果、休眠打破に重要とみられる遺伝子を網羅的に見つけ出し、中でもヒストンH3遺伝子の変動が極めて重要な役割を果たすことを明らかにしました。本研究により、今後様々な細胞が休眠から増殖を始めるメカニズムの解明につながり、新たな制がん標的遺伝子の特定や、がん細胞の増殖を抑える創薬への道筋が期待されます。
本研究成果は、2020年3月9日(月)午前10時(英国時間)に「Nature Communications」のオンライン版に掲載されました。
(1)これまでの研究でわかっていたこと
なぜ、細胞の目覚めを研究するのか?
動物では本来なら休眠状態※4であるべき細胞が目覚めて増殖を開始すると、がん細胞として害をもたらします。また、植物は種子として長期休眠していますが、環境の変化に応じて発芽し、植物体を作り出します。このように細胞が目覚めることは、生物に広く共通した細胞の「運命決定」であり、そのメカニズムを解明することは生物学的に重要であるのみならず、植物環境に関するグリーンイノベーション・制がん創薬研究の観点からも研究が重視されます。
しかし、どのような分子が細胞の目覚めを引き起こすのか、メカニズムはほとんどわかっておらず、今回モデル生物として採用した分裂酵母においても全く知られていません。研究が進まない背景には、後述するように技術的な壁があることが挙げられます。世界初の「目覚め」のメカニズム解明のためには、この壁を乗り越える独自性のある実験アイデアや技術的進歩がなければ達成できないと言えます。
既存の実験手法にみられた技術的限界
本研究グループは細胞の目覚めのメカニズム解明に取り組むために、格好のモデル生物である分裂酵母(学名 S. pombe)に注目しました。分裂酵母は、栄養がなくなると配偶子(胞子※5)を形成しますが、胞子は細胞活動をほとんどおこなわず長期の間休眠した状態を保ちます。そして再び栄養が供給されると、環境の変化を認知して胞子は目覚めます(概要図)。このような休眠打破の現象は「発芽」と呼ばれます※5。

概要図:長期休眠から目覚めるメカニズムの解明
〜シングルセル解析から見えてきた、ヒストンの調節による大規模な遺伝子活性化システムの存在〜
この分子機構を解明するために最も効果的なアプローチは、発芽の過程でどのような遺伝子が発現する(RNA※6に転写される)のかを網羅的に解析する方法だと考えます。これは既存の実験手法では、多数の細胞を集めてRNAを単離することが必要です。しかし胞子細胞は、発芽のタイミングに個体差が大きいために、発芽中の胞子を大量に集めることは不可能でした。したがって、発芽すなわち目覚め始めた細胞内でどのような遺伝子が発現しているのかを調べることは、従来の実験方法では技術的に不可能でした。
(2)今回の研究で新たに実現しようとしたこと、明らかになったこと
技術的限界を乗り越えるにはどうすればよいか?
そこで本研究グループはシングルセル解析技術を改良し、多くの細胞を集めなくても胞子1細胞さえあればRNAを効率的に単離でき、その配列を解読する胞子シングルセルRNA-seq解析※2の手法を開発しました。
次に、独自に開発したシングルセルRNA-seq解析の技法をもとに、目覚め始めた胞子1個1個からRNAを取り出して、分裂酵母全7,000遺伝子の発現状態を解析しました。これを繰り返し、合計64個の「目覚めつつある」胞子における全7,000遺伝子の発現状態をビッグデータとして獲得しました(図1)。

図1:胞子シングルセルRNA-seq解析法を開発し、どの細胞が目覚めの初期段階のものか、仮想時間軸から推定。それらの細胞では、ヒストンH3遺伝子の一つが劇的に発現変動していた。
目覚めの初期段階で劇的に変動する遺伝子は何か?
この「64細胞×7,000遺伝子」のビッグデータから見つけ出したいのは、細胞が長期休眠から目覚め始める際に、どの遺伝子が発現上昇ないし低下するかです。そのような遺伝子の中にこそ、目覚めのメカニズムの鍵を握る遺伝子が存在すると考えました。しかしながら、64個の胞子は、見た目はどれも似たような球形の細胞であり、どれが「目覚め始めの初期段階」の細胞で、どれが「すっかり目覚めた後期段階」の細胞であるのか、見た目では判断できませんでした。
そこで、バイオインフォマティクス技術※7を駆使して、64個の遺伝子発現状態がどのように類似しているかを比較解析することで、64個の細胞がどのように「初期→中期→後期」と時間的に推移したのかを仮想的な時間軸(バーチャル・タイムライン)に沿って並べることに成功しました(図1)。すなわち、バーチャル・タイムラインの出発点あたりに位置するいくつかの細胞こそが、64個の中で最も「目覚め始めの初期段階」にあると言えます。
このようにして、目覚め始めの細胞がどれであるのかが明らかになりました。さらにバイオインフォマティクス解析を続け(図1)、その中でも最も劇的に発現が変動しがちな遺伝子として、ヒストンH3遺伝子※8の一つ(ヒストンH3 #1)を発見しました。
ヒストンH3の遺伝子は3つあるが、そのなかの1個は目覚めるための遺伝子であった
ヒストンは8量体としてDNAに巻き付き(ヌクレオソーム構造)、遺伝子DNAの発現状態をコントロールすることが広く知られる因子であり(図2)、ヒストンH3はそのなかの1因子です。分裂酵母にはヒストンH3を作るための遺伝子が3種類存在しており、なぜ同じものが3つも存在するのか、それぞれの遺伝子に異なった役割があるのかどうかなど、全てが謎に包まれていました。
本研究グループは目覚めの初期段階で、ヒストンH3 #1遺伝子の発現が劇的に低下することを見出しましたが(図2)、他の2つのヒストンH3遺伝子(#2と#3)には大きな変動は見られませんでした。そして、ヒストンH3 #1遺伝子を破壊した変異体は、通常の細胞増殖には異常を示しませんでしたが、細胞の発芽が著しく遅れることがわかりました(図2)。
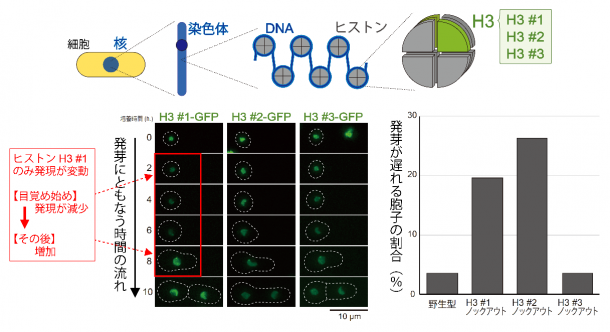
図2 :細胞には3つのヒストンH3遺伝子があるが、そのうちの一つ(H3 #1)は目覚め後に量が変動する。破壊すると目覚めに欠陥を示すため、目覚めに重要な役割を担うことがわかった。
ヒストンH3 #1遺伝子には、その発現量を減少させることで細胞を目覚めさせる重要な働きがあることがわかりました。この遺伝子は細胞増殖時には特別な役割を果たしませんが、胞子から目覚めるときにこそ欠かせない役割を果たす特殊なヒストンH3と推測されます。細胞は目覚めて活動を始めるにあたり、ゲノム全体で莫大な数の遺伝子を活発化させる必要があります。細胞は、それを実現するための秘策として目覚める際にヒストンH3 #1の量を意図的に減少させ、その結果ゲノムのヒストン結合状態を著しく変化させることで、その後のゲノム規模での遺伝子発現を誘導すると考えられます。
(3)そのために新しく開発した手法
研究開始当初には、とても小さな酵母の1細胞(長さおよそ10 µm = 1 mmの100分の1のサイズ)を単離して、その中からRNAを抽出し、さらにその塩基配列※9を解読すること、これら全てにおいて前例がありませんでした。特に大きな課題であったのは酵母が持つ細胞壁の存在で、これが細胞内からRNAを抽出することを困難としました。そこで、本研究グループでは実験手法を開発するところから始めました。この技術開発の成功によって、細胞集団レベルでは捉えることのできなかった、胞子細胞が目覚める分子機構にアプローチが可能となりました。その結果、世界初の「胞子における遺伝子発現プロファイル※2」を作成し、胞子の目覚めのメカニズムに重要な役割を果たすヒストン遺伝子の特定に成功しました。
(4)研究の波及効果や社会的影響
今回のシングルセル発現解析の結果から、休眠打破に重要とみられる遺伝子を網羅的に見つけ出すことに成功しました。今回はヒストンH3遺伝子について解析しましたが、他にも重要な因子が選び出されている可能性が高く、細胞の目覚め研究における「宝の山」となる可能性があります。
休眠と目覚めに関する謎はつきません。細胞は休眠しているにも関わらず、周囲の環境に栄養が供給されたことを認識できるのはなぜか、それがどのようにヒストンH3 遺伝子の発現を変動させるのか、そもそも栄養がなくても死なずに生存を維持できる「休眠状態」とは細胞にとってどのような状態なのかなどが挙げられます。植物の種子では、1000年前につくられた種子でさえも発芽できることが示されています。周囲に栄養がなくても休眠することで長期にわたり生存を維持できる「省エネルギー」と「長寿」の秘訣が隣り合わせで胞子に隠されていると考えます。このことは、動物においては「がん化」という不都合な目覚めのメカニズム解明と予防策の究明に繋がると考えられます。
(5)今後の課題
ヒストンH3遺伝子の制御による染色体のゲノム規模での変化は、受精卵からの発生、植物種子の発芽、がん細胞の増殖再開のメカニズムと類似していると考えられます。ヒストンを人為的に制御すれば、あらゆる生物で細胞の休眠打破を人工的にコントロールすることができると考えます。このように、今後様々な細胞が休眠から増殖を始めるメカニズムの解明を目指して、植物環境におけるグリーンイノベーションの基盤としたり、新たな制がん標的遺伝子の特定や、がん細胞の増殖を抑える創薬につなげたりと、産学連携ベースで研究を進め、SDGsの実現に向けての発展が期待されます。
(6)用語説明
※1 産総研・早大 生体システムビッグデータ解析オープンイノベーションラボラトリ(CBBD-OIL)
2016年7月に早稲田大学と産業技術総合研究所(以下、産総研)が共同で設立した組織。産総研のオープンイノベーションラボラトリ(OIL)は、産総研の第4期中長期計画(平成27年度~31年度)で掲げている「橋渡し」を推進していくための新たな研究組織の形態で、CBBD-OILはその第4号案件。また、CBBD-OILは産総研が私立大学と共同で設立した初めての組織である。
※2 シングルセル解析技術(シングルセルRNA-seq解析)、遺伝子発現プロファイル
遺伝子が働くときにはDNAからRNAが転写されるが、数千ある遺伝子は全てが均等にRNAになるわけではなく、その場面で個々の遺伝子のRNA量は異なる。1細胞から全RNAを採取し、各遺伝子のRNA発現量を調べて一覧を作る(=プロファイルを作成する)ことで、各遺伝子がどれだけ働いているかがわかる。
※3 分裂酵母
ヒトと同じ真核生物に分類される単細胞の生物。ヒト細胞でも見られる特徴を多く持ち、その仕組みが単純でわかりやすく、遺伝子改変をしやすいことから、生物学ではモデル生物としてよく使われる。
※4 休眠状態
細胞が代謝を極限まで抑えることで細胞活動を停止している状態。周囲の環境が生育に適していないと細胞は休眠状態に突入する。これは細胞が劣悪な環境に耐えて長期生存をはかる戦略だと考えられる。休眠状態の細胞は環境ストレスに強い耐性があり、長い間休眠状態を維持し続けることができる。
※5 胞子、胞子の発芽
胞子は分裂酵母の休眠状態のこと。分裂酵母は周囲の栄養がなくなると雌雄の細胞が融合し、次の世代となる配偶子すなわち胞子が作られる。胞子は長い間休眠して生存可能であるが、再び環境中に栄養が供給されると、休眠から目覚めて増殖を開始する。この休眠から目覚める過程を「発芽」という。
※6 RNA
DNAがもつ遺伝情報をコピーした物質。リボ核酸 (ribonucleic acid) の略。一つの遺伝子から複数のRNAが作られ、その量によって遺伝子の働く強さが決まる。
※7 バイオインフォマティクス技術
生命科学と情報科学とを融合した技術。近年、生命科学の分野では、大量の実験データを一度に獲得できる手法が発展しており、そこから特に重要な情報を収集するためには情報科学の技術が必要不可欠である。
※8 ヒストン、ヒストンH3
ヒストンは細胞内でDNAに巻き付くタンパク質の複合体。DNAを小さく折りたたむことで核の中に収納している。また、結合するDNA上に存在する遺伝子の発現しやすさを決める重要な役割も担う。ヒストンはH2A、H2B、H3、H4と呼ばれる4種類のタンパク質からなり、中でもH3は化学的な制御を受けることで、巻き付くDNAから発現するRNA量の大小を決めるため、昨今大きな注目を集めている。
※9 塩基配列
DNAやRNAを構成する塩基と呼ばれる物質の順番を示したもの。塩基にはアデニン(A)、チミン(T)、グアニン(G)、シトシン(C)、ウラシル(U)の5種類がある。DNAはA、T、G、Cの4種類、RNAはA、U、G、Cの4種類からなり、それぞれが直鎖上に並んだ構造をしている。塩基がどのような順番で並んでいるかによって、その遺伝子の機能が決まる。
(7)論文情報
雑誌名:Nature Communications
論文名:Time-lapse single-cell transcriptomics reveals modulation of histone H3 for dormancy breaking in fission yeast
執筆者名(所属機関名):露崎隼1,2、細川正人3,4、有川浩司2,3、依田卓也1、岡田直幸1,5、竹山春子1,2,3,4、佐藤政充1,4,6
(1.早稲田大学 大学院先進理工学研究科 生命医科学専攻、2.産業技術総合研究所 産総研・早大 生体システムビッグデータ解析オープンイノベーションラボラトリ (CBBD-OIL)、3.早稲田大学 ナノ・ライフ創新研究機構、4.早稲田大学 先進生命動態研究所、5.Instituto de Biologia Molecular e Celular, Instituto de Investigacão e Inovacão em Saude (i3S), Universidade do Porto、6.早稲田大学 構造生物・創薬研究所)
掲載日時(英国時間):2020年3月9日 午前10時
掲載日時(日本時間):2020年3月9日 午後7時
DOI:https://doi.org/10.1038/s41467-020-15060-y
(8)研究支援
研究費名:文部科学省 新学術領域研究
研究課題名:シングルセル発現解析と核膜変異体ライブラリを用いた転写サイクル始動機構の解明
研究代表者名(所属機関名):佐藤 政充(早稲田大学 理工学術院)
なお、本研究の一部は、国立研究開発法人 日本医療研究開発機構(AMED)の創薬等先端技術支援基盤プラットフォーム 課題番号 JP19am0101104による支援を受けておこないました。







