化合物の標的分子の同定を効率化
新しいshRNAスクリーニングで複数試料の同時解析を可能に
理化学研究所(理研)環境資源科学研究センターケミカルゲノミクス研究グループの髙瀬翔平研修生、吉田稔グループディレクター、松本健専任研究員、早稲田大学理工学術院の新井大祐次席研究員、中尾洋一教授らの共同研究グループ※は、薬の元となる化合物の作用メカニズムの同定に利用される「shRNA [1]ライブラリースクリーニング[2]」を効率化し、この手法を用いてアポトーシス[3]を誘導する海洋天然物オーリライドB[4]の作用メカニズムに関わる新たな遺伝子を同定しました。
培養細胞にshRNAを導入することで、特定の遺伝子をノックダウン[5](タンパク質の合成を抑制)することができます。さまざまなshRNAを集めたものをshRNAライブラリーと呼びますが、培養細胞群にshRNAライブラリーを導入することで、さまざまな遺伝子をノックダウンさせた培養細胞群を作成することができます。このshRNAライブラリーを導入した培養細胞群に対して化合物処理を行うことで、化合物の作用メカニズムに関わる遺伝子を網羅的に調べること(スクリーニング)ができます。このshRNAライブラリースクリーニングは、化合物の直接の標的分子の同定だけでなく、化合物への感受性に関わる遺伝子群を同定できる点で優れています。しかしこれまで、細胞でのスクリーニング後のshRNA同定の段階で、サンプルごとに次世代シークエンサー[6]による解析を行っていたため、処理効率が低くコストが高いという課題がありました。
今回、共同研究グループはゲノムワイドなshRNAライブラリースクリーニングにおいて、次世代シークエンサーで一度に複数のサンプルを解析できる方法を確立し、再現性や定量性について確認しました。これにより、処理効率の向上と低コスト化(従来の数分の1)が実現されました。次に、この方法を用いてアポトーシスを誘導する海洋天然物のオーリライドBの作用メカニズムに関わる遺伝子の同定を行いました。その結果、オーリライドBへの感受性に関わる遺伝子としてATP1A1遺伝子を同定しました。ATP1A1遺伝子は、細胞膜のNa+/K+ATPアーゼ[7](ナトリウムポンプ)のαサブユニットの遺伝子です。また、ATP1A1タンパク質の阻害剤である強心配糖体[8]のウアバイン[9]をオーリライドBと共に細胞に作用させたところ、単独の場合よりも低濃度でオーリライドBが細胞増殖抑制を引き起こすことが分かりました。
今回確立した手法により、今後、多くの化合物の作用メカニズムに関わる遺伝子を明らかにできると期待できます。
本研究は、英国のオンライン科学雑誌『Scientific Reports』(5月17日付け:日本時間5月17日)に掲載されました。
※共同研究グループ
- 理化学研究所 環境資源科学研究センター ケミカルゲノミクス研究グループ
研修生 髙瀬 翔平 (たかせ しょうへい)
グループディレクター 吉田 稔 (よしだ みのる)
専任研究員 松本 健 (まつもと けん)
テクニカルスタッフⅠ 黒川 留美 (くろかわ るみ)
- 明治大学大学院 農学研究科
准教授 久城 哲夫 (くしろ てつお)
- 早稲田大学理工学術院
教授 中尾 洋一 (なかお よういち)
次席研究員 新井 大祐 (あらい だいすけ)
- 北海道大学大学院 地球環境科学研究院
教授 沖野 龍文 (おきの たつふみ)
- ミクロネシア連邦チューク州コミュニティ カレッジ
Campus Dean カインド・カネモト・カント(Kind Kanemoto Kanto)
1.背景
培養細胞にshRNAを導入することで、特定の遺伝子をノックダウン(タンパク質の合成を抑制)することができます。さまざまなshRNAを集めたものをshRNAライブラリーと呼びますが、培養細胞群にshRNAライブラリーを導入することで、さまざまな遺伝子をノックダウンした培養細胞群を作成することができます。このshRNAライブラリーを導入した培養細胞群に対して化合物処理を行うことで、化合物の作用メカニズムに関わる遺伝子を網羅的に調べること(スクリーニング)ができます(図1)。
例えば、shRNAライブラリーを導入した培養細胞群に、細胞増殖に影響を与える化合物を処理し、生き残った細胞がどのshRNAを発現していたかを非処理細胞(対照)と比較します。該当するshRNAを同定することで、細胞増殖に影響を与える化合物の作用メカニズムに関わる遺伝子だけでなく、化合物と合成致死性[10]を示す遺伝子の情報を得ることができます。しかし、これまで共同研究グループが用いているshRNAライブラリーでは、shRNAの同定の際、サンプルごとに次世代シークエンサーによる解析を行っていたため、処理効率が低くコストが高いという課題がありました。
そこで、共同研究グループは、shRNAの同定に複数サンプルを同時に解析できる手法の開発を試みました。
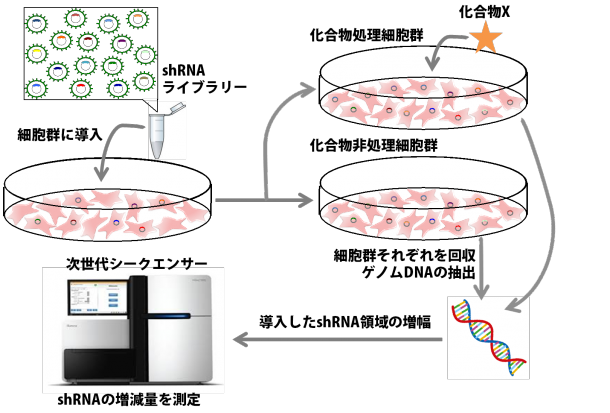
図1 shRNAライブラリースクリーニングの概要図 さまざまなshRNAを集めたshRNAライブラリーを培養細胞群に導入することで、さまざまな遺伝子をノックダウンした培養細胞群を作成することができる。shRNAライブラリーを導入した後に化合物で処理した細胞群と非処理群のshRNAの発現状況を比較することで、化合物の作用メカニズムに関わる遺伝子を網羅的に調べること(スクリーニング)ができる。
2.研究手法と成果
共同研究グループはまず、次世代シークエンサーによる解析の前に、異なるサンプル同士を区別できるよう、8種類のインデックス配列(8塩基配列)を持つプライマー[11]を用いてshRNAの検出用特異的配列であるバーコード部分(18塩基配列)をポリメラーゼ連鎖反応(PCR)[11]で増幅しました。これにより、いくつかのサンプルのPCR産物を混合してシークエンス解析を行った後に、インデックス配列によってサンプルを分類し、サンプル内でのshRNAの増減を調べることが可能になりました(インデックス付きシークエンス法、図2)。
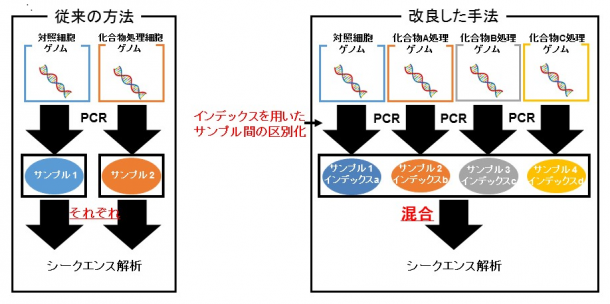
図2 インデックス付きシークエンス法 従来法(左)ではサンプルごとに別々にシークエンス解析をしなければならないが、インデックス付きシークエンス法(右)では、ポリメラーゼ連鎖反応(PCR)を行う際にインデックス配列を付けることにより、シークエンス解析後に複数のサンプルを区別できるようになった。
次に、同一のサンプルをこのインデックス付きシークエンス法と従来法でシークエンス解析を行い比較したところ、インデックスを付けても従来法に比べて結果に影響がないことが分かりました。さらに、同一のサンプルを異なるインデックス配列を持つ2種類のPCRプライマーで増幅しシークエンス解析を行っても、非常に再現性の高い結果が得られました(図3)。

図3 インデックス付きシークエンス法の再現性 同じサンプルから、2種類のインデックス付きプライマーを用いてPCRを行い、それぞれのバーコードのリード数を比較した。2種類のインデックス間(X軸:インデックスa、Y軸:インデックスb)で非常に再現性の高い結果が得られた。
また、本来、shRNAライブラリー中にないバーコード配列を添加してインデックス付きシークエンス法での解析を行ったところ、添加量に伴ってシークエンスのリード数が増加したことから、本法が定量性よくshRNA量を決定できることが分かりました。また、解析コストも従来の数分の1になりました。
そこで実際に、このshRNAライブラリースクリーニングで化合物の標的分子が同定できるかどうかを、DNAトポイソメラーゼⅡ[12]の阻害剤で抗がん剤として使用されるエトポシドを用いて調べました。その結果、エトポシド処理細胞中のDNAトポイソメラーゼⅡのmRNAに対応するshRNAが非処理細胞に比べて著しく増加したことから、本法によって化合物の標的分子の同定が可能であることが確かめられました。
次に、ラン藻から単離されアポトーシス(プログラムされた細胞死)誘導活性を持つ、海洋天然物のオーリライドBについて、shRNAライブラリースクリーニングを行いました。非処理細胞に比べてオーリライドB処理細胞で減少するshRNAに注目して解析を進め、候補遺伝子に対するsiRNA[1]で二次スクリーニングを行いました。その結果、「ATP1A1遺伝子」のノックダウンによって、細胞のオーリライドBに対する感受性が非常に高くなる、すなわち低濃度のオーリライドBで細胞増殖抑制が引き起こされることが分かりました(図4)。
ATP1A1は、細胞膜のNa+/K+ATPアーゼ(ナトリウムポンプ)の酵素活性を担うαサブユニットの遺伝子です。Na+/K+ATPアーゼのαサブユニットは、強心作用を持つ強心配糖体によって阻害されることが知られています。そこで、強心配糖体の一つであるウアバインとオーリライドBで細胞を共処理したところ、オーリライドB単独の場合よりも低濃度でオーリライドBが細胞増殖抑制を引き起こしました。これにより、ウアバイン存在下では、細胞のオーリライドBに対する感受性が高まることが分かりました(図4)。
さらに、オーリライドBによるアポトーシス誘導の指標であるPARP(ポリ(ADPリボース)ポリメラーゼ1)[13]の切断が、ウアバインとの共処理によって促進されることが分かりました。以上の結果は、ATP1A1タンパク質が細胞のオーリライドBに対する感受性を決める因子であることを示しています。
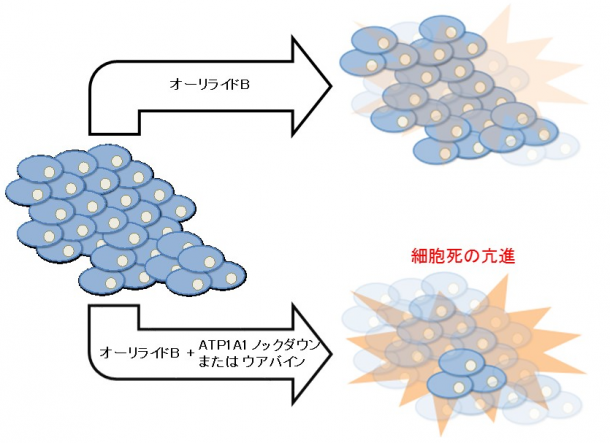
図4オーリライドBとATP1A1阻害によるアポトーシス誘導の模式図 ATP1A1のノックダウン、あるいは、ATP1A1阻害剤であるウアバインとの併用によって、オーリライドBによる細胞死(アポトーシス)が亢進する。
3.今後の期待
今回開発したインデックス付きシークエンス法によってshRNAライブラリースクリーニングの処理効率を上げることができるため、ヒト培養細胞を使って遺伝学的に化合物の標的経路や合成致死経路の同定が進むと期待できます。
shRNAやgRNA[14]ライブラリーでのスクリーニングは化合物の標的同定以外にも、がん細胞の脆弱性の要因の同定や、疾患モデル細胞のプロファイリングを通じた新たな治療標的分子の同定などにも利用できるため、今回の方法は今後、幅広く応用可能な技術といえます。
白血病、腎臓がん、前立腺がんの細胞株において、オーリライドBの感受性が高いことが知られています。一方、強心配糖体もさまざまながん細胞に対して選択的に増殖抑制活性を持つことが分かっています。今後、オーリライドBと強心配糖体の併用により、どのようながん細胞に対する細胞増殖抑制効果が強いのかなどを明らかにすることによって、新たな治療戦略につながる可能性があります。
4.論文情報
- タイトル:A quantitative shRNA screen identifies ATP1A1 as a gene that regulates cytotoxicity by aurilide B
- 著者名:Shohei Takase, Rumi Kurokawa, Daisuke Arai, Kind Kanemoto Kanto, Tatsufumi Okino, Yoichi Nakao, Tetsuo Kushiro, Minoru Yoshida, and Ken Matsumoto
- 雑誌:Scientific Reports
- DOI:10.1038/s41598-017-02016-4
5.補足説明
- [1] shRNA、siRNA:shRNA(short hairpin RNA)はヘアピン型の小さなRNA(およそ60塩基配列)であり、細胞内のRNA切断酵素で切断されることによって、21~23塩基のsiRNA(small interfering RNA)となる。shRNAの配列を選んで細胞内に存在させると、siRNAがRNA分解酵素を特定のメッセンジャーRNA(mRNA)に作用させるため、特定の遺伝子の発現(タンパク質の合成)を特異的に阻害すること(RNA干渉)ができる。
- [2] shRNAライブラリースクリーニング:細胞内に存在するさまざまなmRNAに対応するshRNAを集めたものを、shRNAライブラリーと呼ぶ。shRNAライブラリーを細胞に導入することで、それぞれのmRNAから合成されるタンパク質の機能を網羅的に調べる方法をshRNAライブラリースクリーニングという。shRNAライブラリーには、さまざまなmRNAに対するshRNAが混合されたプール型と、混合されずに別々の状態になっているアレイ型があり、今回の研究ではプール型を用いた。
- [3] アポトーシス:プログラムされた細胞死を指し、生理的には個体の正常な発生に必要であり、またがん化した細胞などの除去の目的でも積極的に引き起こされる。
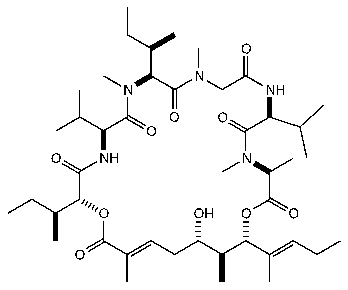
- [4] オーリライドB:ラン藻から単離された海洋天然物で、がん細胞の培養液に添加すると細胞増殖を抑制する活性を持つ。構造は下図の通り。
- [5] ノックダウン:遺伝子の機能を大きく減衰させるが、完全には欠失させない状態にすること。mRNAの分解促進や、タンパク質の翻訳阻害など、遺伝子破壊以外の操作により行う場合が多い。
- [6] 次世代シークエンサー:DNAの塩基配列を決定するための装置で、従来の方法と異なる方法でより高速高精度に塩基配列を決定することができる。ヒトゲノム解読を目指して開発された。
- [7] Na+/K+ATPアーゼ:細胞内は細胞外に比べるとナトリウムイオン(Na+)濃度が低く、カリウムイオン(K+)が高い。この状態を保つために、ナトリウムイオン(Na+)を細胞外へ、カリウムイオン(K+)を細胞内へ、ATPを消費して輸送するポンプ膜タンパク質がNa+/K+ATPアーゼである。ナトリウムポンプとも呼ばれ、αとβサブユニット、補足的なγサブユニットからなる。
- [8] 強心配糖体:強心作用(心臓の収縮力を強くする)を持つステロイド配糖体のことで、心不全や不整脈用の薬として用いられる。Na+/K+ATPアーゼに特異的に結合して活性を阻害する。
- [9] ウアバイン:強心配糖体の一つで、リンドウ目キョウチクトウ科の植物Strophanthus gratusなどの種子に含まれる。アフリカのソマリ族の間で、毒矢の毒として用いられていた。
- [10] 合成致死性:単独の変異では細胞が死に至らない複数の遺伝子が同時に変異することによって、細胞が死に至る関係性のこと。
- [11] プライマー、ポリメラーゼ連鎖反応(PCR):微量DNAを酵素反応で増幅する反応をPCRと呼ぶ。プライマーはPCRの際に用いられる2種類のオリゴヌクレオチド。
- [12] DNAトポイソメラーゼⅡ:二本鎖DNAは二重らせん構造を形成している。転写、複製、修復の際には二重らせん構造をほどく必要があるが、その際に問題になるねじれを調節しているのがトポイソメラーゼである。DNAの1本だけを切断する酵素をトポイソメラーゼⅠ、2本とも切断する酵素をトポイソメラーゼⅡという。
- [13] PARP(ポリ(ADPリボース)ポリメラーゼ1):タンパク質をポリADPリボシル化する酵素。細胞のアポトーシスの際にタンパク質分解酵素によって切断されることが知られており、アポトーシスが起きている細胞の指標として用いられる。
- [14] gRNA:gRNA(guide RNA)は小さいRNAで、ゲノムDNAの特定の部分に相補的な配列を含む。CRISPR-Casシステムによるゲノム編集において、Cas9ヌクレアーゼをゲノムDNAの特定の部分に作用させるために用いられる。







