多種多様な細菌のゲノム情報を一挙に個別解読
ヒトの健康と関わる腸内細菌叢の機能理解を助け、医療分野に貢献
発表のポイント
- 多種多様な細菌の集合から、細菌1細胞ごとに網羅的にゲノムを解読する技術を開発した。
- 網羅的に収集した腸内細菌ゲノムから、腸内環境で食物繊維の代謝を行う細菌・遺伝子を特定した。
- 本法は細菌ゲノムを圧倒的速度で収集して細菌叢の機能理解につなげ、医療・産業への応用に貢献する。
早稲田大学理工学術院の細川正人(ほそかわまさひと)次席研究員(研究院講師)、竹山春子(たけやまはるこ)教授らの研究グループは、多種多様な細菌から1細胞ごとにゲノム(※1)を解読する技術「SAG-gel」を開発しました。これにより、細菌の集合体である腸内細菌叢の中から、個々の細菌ゲノム情報を収集し、ホストに影響を与えうる細菌や遺伝子を探索することが可能となりました(図1)。
現在、ヒトの健康・疾患状態と腸内細菌叢の関係に注目した研究が世界各地で進められていますが、その分析には様々な細菌が混在したゲノムDNAを用いる方法が一般的となっています。しかし、混在した塩基配列を正しく整理して理解することは難しく、特定の機能を持つ細菌・遺伝子を探索するなどの応用が困難でした。
SAG-gelは、微小なカプセル内に細菌叢を物理的に1細胞ごとに分離し、個別の反応環境を構築してからゲノム解読処理をスタートします。得られるすべての塩基配列情報が1細胞由来であるため、複雑な計算処理を必要とせずに短時間でドラフトゲノム(全ゲノム配列の概要)を決定でき、収集したデータから標的の機能を持つ細菌・遺伝子を探索することが可能です。本法により、環境細菌やヒト腸内細菌などの微生物ゲノム情報を圧倒的な速度で収集することが可能となります。今後獲得される細菌ゲノム情報は、有用な酵素や抗生物質などの探索用の情報資源として活用できるほか、ヒトの健康と関わる細菌叢の機能理解のための基礎情報として、産業応用や医療分野への貢献が期待されます。
本研究成果は、『Microbiome』に2020年1月23日(現地時間)に掲載されました。
(1)これまでの研究でわかっていたこと
現在、ヒトや動物の健康・疾患状態と腸内細菌叢の関係に注目した研究が世界各地で進められています。腸内細菌叢は多種多様な細菌の集合体ですが、その機能や多能性の詳細には多くの謎が残されています。現在、研究によく用いられているメタゲノム解析(※2)法では、細菌叢から、多種の細菌の遺伝情報が混在したDNAを抽出し、遺伝子の一部を増幅したものや、全体を断片化したものから、その塩基配列を読み取ります。この雑多な塩基配列を情報処理によって類似度などを指標に分別したのち、公共のデータベースに照合することで、細菌の種や遺伝子の存在を推定します。しかし、この方法では、照合元となる微生物ゲノム(リファレンスゲノム)情報が充実していないこと、混在する類縁菌を情報処理によって区別することが困難であることなどが課題となっています。今後、個人の腸内細菌叢の特徴を解像度よく捉えることを実現するには、リファレンスゲノムを拡充させるとともに、個々の細菌のゲノムを正確かつ網羅的に決定する方法が必要とされています。
(2)今回の研究で新たに実現しようとしたこと、明らかになったこと
本研究では、多様で複雑な細菌叢から個々の細菌ゲノムを決定するために、細胞1つからゲノム配列を解読するシングルセルゲノム解析(※3)技術「SAG-gel(Single-cell Amplified Genome in gel)」の開発に取り組みました。本法では、はじめに細菌叢を物理的に1細胞ごとに分離してから、ゲノム解読処理をスタートします。獲得したすべての塩基配列情報が1細胞由来であるため、従来は数時間から数日要していた複雑な計算処理を必要とせず、10分程度の短時間の計算でドラフトゲノム(全ゲノム配列の概要)を獲得することができます。サンプル中の存在比が小さい細菌種からゲノム情報を取得することもでき、類縁菌間での細かな塩基配列の違いまでを明らかにする特徴を持ちます。
本法の応用として、プレバイオティクス(※4)として利用される食物繊維イヌリンを利用する腸内細菌・遺伝子を探索しました。マウスにイヌリンを2週間給餌したのち、糞便中の腸内細菌叢をSAG-gelを用いて解析して300個超の未培養細菌のゲノム配列情報を一挙に獲得しました(図1)。得られた高品質のゲノム情報から、イヌリン利用に必要な遺伝子を探索し、腸内細菌が産生しホストに影響を与えることで知られる短鎖脂肪酸に関わる遺伝子の有無を調べました。この結果、イヌリンを栄養源として腸内で優先的に増殖する応答者であり、特定の短鎖脂肪酸を高産生する細菌を特定することができました(図2)。
(3)そのために新しく開発した手法
従来の微生物対応シングルセルゲノム解析技術は、非常に微量な1細胞ゲノムDNAを扱う多段階反応は困難で、反応が不十分なため精度・スループットが低く、一部のゲノム情報しか得られませんでした。当研究グループは微生物のシングルセルゲノム解析技術を長年開発し、特にマイクロ流路を用いて1細胞ゲノムDNAを扱う技術を開発してきましたが、本研究ではマイクロ流路を介して作られる微小なゲル状のカプセルを新しく利用することで、さらに解析の効率・精度が向上したシングルセルゲノム解析技術「SAG-gel」を確立しました。本法では、マイクロ流路内で細菌が1細胞ずつ小さなゲル状のカプセルに高速に封入され、細胞1つ1つが小さな反応環境に分離されます。秒間数百個もの速度でカプセルが生成され、総計で数万~数十万個ものシングルセル封入カプセルが作られます。この無数のゲルカプセルは、1つの試験管にまとめて回収され、各種溶剤に順次浸漬されることで、カプセル内の細菌の溶解、DNAの精製・増幅が連続的に行われます(図1a)。この増幅ゲノムを並列的に読み取り、配列を解析することで、各細菌のゲノム情報が一挙に獲得されます。
本法のポイントはゲルカプセルを反応環境として新しく利用したことです。カプセルは外部からの反応環境汚染を抑制するほか、強力な溶剤を用いた細菌溶解と夾雑物洗浄除去操作の支持体として役立ち、これまで不可能であった1細胞由来ゲノムDNAの精製・反応などの連続多段階の処理を実現しました。カプセル内で十分にDNAを精製したのちにゲノム増幅に移行するため、多様な細菌への対応とゲノム解読の効率の改善が実現しています。また、処理操作の複雑性も解消されており、1つの試験管に浮遊したゲルカプセルに対し、試薬を順次添加・置換する簡易な操作で、数十万の細菌1細胞の超並列ゲノム増幅を実行することができます。
(4)研究の波及効果や社会的影響
これまでの未培養細菌を対象としたシングルセルゲノム解析技術は、非常に煩雑で非効率的な手順が取られており、精度・速度面で実用的な技術がありませんでした。SAG-gelは、幅広い微生物種に適応可能で、一般実験室でも実行できるだけでなく、高精度なゲノム情報を一度にたくさん得ることが可能です。本法を活用すれば、海洋や土壌などに生息する環境細菌やヒト腸内細菌などの微生物ゲノム情報を圧倒的な速度で収集できます。この情報は、リファレンスゲノムとして、様々な微生物研究でのデータベースとして活用されます。例えば、生物遺伝情報資源として、細菌の作る酵素や抗生物質の探索に利用できるほか、ヒトの健康と関わる細菌叢の機能理解のための基礎情報として、産業応用や医療分野へ貢献します。
細川次席研究員(研究院講師)は、本法を社会実装するために、早稲田大学関係ベンチャー「bitBiome株式会社」を2018年に創業し、SAG-gelを活用したシングルセルゲノム解析サービスを展開しています。
(5)今後の課題
今回の結果により、未培養細菌のゲノム情報を一挙に獲得できるようになりましたが、ゲノム情報には読み取りが難しい領域が存在し、これらはギャップとして残されています。将来は、これらのギャップを埋めて読み取り領域を増やすことが必要であり、遺伝子領域の機能推定精度を向上させることが必要です。また、本法を活用してヒトの腸内細菌叢や様々な環境細菌を対象とした実証試験を行う予定です。
(6)用語解説
- ※1 ゲノム
遺伝情報の全体・総体を意味する。本研究では、特に細菌のDNA上の全遺伝情報を指しており、ゲノム解析はDNA塩基配列を解読することを指す。
- ※2 メタゲノム解析
細菌叢から分離・培養を経ずに抽出したゲノムDNAをもとに、DNA塩基配列を解読する手法。得られた塩基配列から、細菌叢に存在する各種細菌の系統組成と遺伝子機能組成などを調べることに優れる。
- ※3 シングルセルゲノム解析
細菌1細胞から抽出したゲノムDNAをもとに、増幅などの前処理を行い、DNA塩基配列を解読する手法。得られた塩基配列から、各細菌のドラフトゲノム(ゲノム配列の概要)を得ることに優れる。
- ※4 プレバイオティクス
消化管上部で分解・吸収されず、大腸に共生する腸内微生物の選択的な栄養源となり、それらの増殖を促進し、腸内細菌叢のバランスを改善し、ヒトの健康増進・維持に役立つ食品成分を指す。
(7)論文情報
- 雑誌名:Microbiome
- 論文名:Single-cell genomics of uncultured bacteria reveals dietary fiber responders in the mouse gut microbiota
- 執筆者名(所属機関名):Rieka Chijiiwa†(早大・産総研CBBD-OIL), Masahito Hosokawa*,†(早大・JSTさきがけ), Masato Kogawa (早大・産総研CBBD-OIL), Yohei Nishikawa (早大・産総研CBBD-OIL), Keigo Ide (早大・産総研CBBD-OIL), Chikako Sakanashi (早大), Kai Takahashi (早大), Haruko Takeyama*(早大・産総研CBBD-OIL) † These authors equally contributed to this work. * Corresponding authors
- 掲載予定日(現地時間):2020年1月23日
- DOI:10.1186/s40168-019-0779-2
(8)参考図
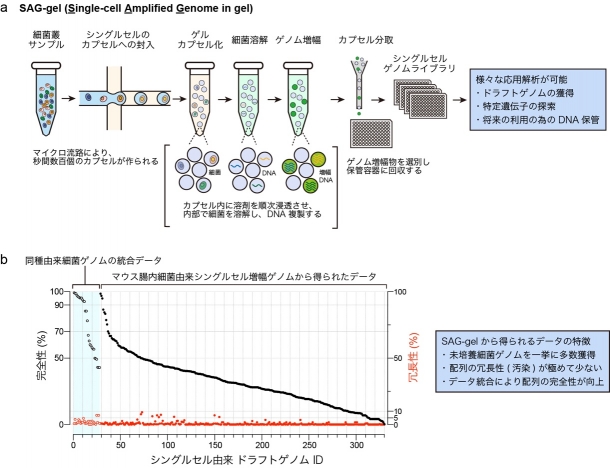
図1(a) 微生物の網羅的シングルセルゲノム解析を可能とするSAG-gel、(b)マウス糞便中細菌から解析したシングルセルゲノムの品質評価(300個を超える細菌ゲノムを取得) 黒:シングルセルから解読したゲノムの完全性 (50%以上であれば中品質ドラフトゲノムと評価される)、赤:シングルセルから解読したゲノムの冗長性 (大きいほどデータが汚染されていることを示す。10%以下であることが望ましい)。重複する細菌種のデータを統合することで、データ品質が改善する(水色部) 。

図2 (a) Bacteroides属細菌のゲノム上にコードされたイヌリン利用遺伝子座の比較、(b)新規獲得したイヌリン利用細菌のドラフトゲノムから予測したイヌリン取り込みタンパク質の構造図
(9)研究助成
- 研究費名:基盤研究(S)
- 研究課題名:新規生理活性物質生産株の超ハイスループットスクリーニングプラットフォーム構築
- 研究代表者名(所属機関名):竹山春子(早大) 研究分担者名(所属機関名):細川正人(早大)
- 研究費名:基盤研究(B)
- 研究課題名:1細胞内のゲノム構造と転写活性制御を紐解くイメージ・シーケンス統合解析
- 研究代表者名(所属機関名):細川正人(早大)
- 研究費名:科学技術振興機構 戦略的創造研究推進事業さきがけ
- 研究課題名:組織内の細胞多様性を明らかにする超並列ゲノム解析技術の創成
- 研究代表者名(所属機関名):細川正人(早大)
- 研究費名:科学技術振興機構 戦略的創造研究推進事業CREST
- 研究課題名:シングルセルゲノム情報に基づいた海洋難培養微生物メタオミックス解析による環境リスク数理モデルの構築
- 研究代表者名(所属機関名):竹山春子(早大)







